Alexis Mendoza-León, PhD. Email: [email protected]
La coexistencia entre un organismo patógeno y su hospedero, no es más que un proceso coevolutivo, donde cada organismo trata de sobrevivir estableciendo mecanismos apropiados de defensa o evasión. Un ejemplo de ello es la interacción virus-célula (eucariota o procariota), ej. virus-bacteria, donde esta última constantemente se encuentra expuesta a la infección viral, que no es más que la inyección del material genético del virus y el aprovechamiento de la célula maquinaria celular para multiplicarse y producir nuevos virus. En respuesta, la célula bacteriana ha desarrollado un arsenal de mecanismos de defensa para contrarrestar la acción viral, como por ejemplo la modificación de receptores de superficie, sistema de modificación-restricción, mecanismos de inhibición de la expresión genética mediados por RNA de interferencia (iRNA), o adquisición y posterior rearreglos de secuencias de DNA por transferencia genética horizontal, entre otros. De igual forma, los virus han desarrollado respuestas de sabotaje y resistencia para evadir estos sistemas celulares antivirales.1,2
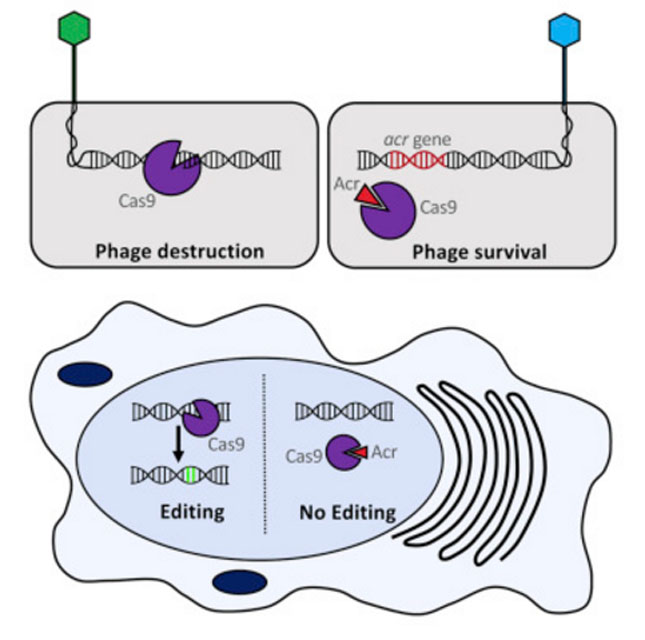
Uno de los mecanismos de inmunidad adaptativa bacteriana que ha recibido gran atención por su especificidad y utilidad en la edición de secuencias del genoma de diferentes organismos, es el sistema CRISPR-Cas (Clustered Regularly Interspaced Short Palindromic Repeat-associated protein; Repeticiones palindrómicas cortas interespaciadas y agrupadas regularmente). El funcionamiento del sistema está definido por tres etapas, en primer lugar, la incorporación en el genoma bacteriano de pequeñas secuencias de DNA provenientes de elementos móviles como plásmidos y virus que las infectan, generándose el locus CRISPR. Segundo, la maquinaria bacteriana transcribe y procesa el locus CRISPR para generar un conjunto de pequeños RNA guías (gcrRNAs), cada uno complementario a las diferentes secuencias previamente incorporadas, región proto-espaciadora. Finalmente, estos gcrRNAs se asocian a proteínas con actividad de nucleasas (tijeras moleculares Cas) formando un complejo ribo-nucleoproteíco, y mediante reconocimiento por complementariedad de bases y la activación enzimática de las tijeras moleculares Cas ocurre la eliminación especifica del DNA invasor, ej. DNA viral infectante. 1,2
A pesar de la alta presión selectiva en el proceso de infección viral por parte de un sistema como CRISPR-Cas y sus variantes (Tipo I, II y III), los virus no han sido erradicados, sugiriendo la coevolución de mecanismos virales de respuesta anti CRISPR (ACr) que suprimen el sistema inmune bacteriano (Viral suppressor CRISPR:VSC), permitiendo la infección viral. La evasión del sistema CRISPR por parte de los virus puede ocurrir por: a. mutación, cambios puntuales o eliminación (deleción) parcial de nucleótidos, en la región proto-espaciadora o de regiones adyacentes, evitando el reconocimiento de la secuencia blanco por parte del guía crRNA; b. presencia de genes cuya expresión son proteínas anti-CRISPR que interfieren en la formación del complejo ribo-nucleoproteíco crRNA-Cas; y c. a través de un sistema viral CRISPR-Cas cuya expresión interfiere con el sistema antiviral de la bacteria. Los primeros ejemplos de proteínas anti-CRISPR producidas por virus se demostraron en Pseudomonas aeruginosa y posteriormente en otras especies bacterianas del phylum Protobacterias y Listeria monocytogenes.3,4
El uso de virus como vehículos de transporte de información genética en terapias genéticas, al igual que su uso potencial en el tratamiento de infecciones bacterianas dado el incremento en la resistencia a antibióticos, determina la importancia de identificar y establecer relaciones de interacción entre estas estrategias anti-virales y las estrategias de resistencia desarrollada por parte de los virus. Uno de los ejemplos que claramente define lo anterior, es la aplicación de CRISPR en el tratamiento de infecciones virales como la de HIV, donde la edición genética esta dirigidas a impedir la multiplicación del virus; trabajos recientes demuestran que el virus HIV desarrolla mutaciones de la secuencia reconocida por el gcrRNA (región proto-espaciadora), previniendo de esta manera la acción del sistema CRISPR y en consecuencia favoreciendo la propagación viral, lo cual limitaría el uso de CRISPR en una posible terapia contra HIV.5 Por otro lado, CRISPR se ha utilizado de manera eficiente en la eliminación de una gran cantidad de retrovirus endógenos integrados en el genoma del cerdo, los cuales habían sido hasta ahora una barrera importante en el uso potencial de estos animales como fuente de órganos para trasplantes en humanos. El monitoreo de la ausencia de potenciales respuestas de defensa por parte de los virus, que pudiesen relacionarse con potenciales infecciones generadas por estos virus, probablemente será determinantes en el uso de estos órganos en el proceso de xenotransplantes. 6
La dinámica del equilibrio entre los mecanismos de defensa antiviral por parte de bacterias y las respuestas de resistencia de virus, influye en la variabilidad genética de ambos organismos. El conocimiento de la relación y dinámica de estos mecanismos celulares de defensa antiviral y las respuestas de resistencia por parte de los virus es de gran importancia
Referencias
- Wiedenheheft B. 2013. In defense of phage. RNA Biology. 10:5, 886-890. http://dx.doi.org/10.4161/rna.23591
- van der Oost J. and Brouns S.J.J. 2015. CRISPR sabotage. Genome Biology 16:248.DOI 10.1186/s13059-015-0820-0
- Pawluk A. et al. 2016. Naturally occurring off-switches for CRISPR-Cas9. Cell 167. http://www.cell.com/cell/fulltext/S0092-8674(16)31589-6 , DOI: 10.1016/j.cell.2016.11.017
- Rauch et al. 2017. Inhibition of CRISPR-Cas9 with Bacteriophage Proteins. Cell 168, 1–9. http://dx.doi.org/10.1016/j.cell.2016.12.009
- Wang et al. 2016. CRISPR/Cas9-derived mutations both inhibit HIV-1 replication and accelerate viral escape. Cell Reports, 2016 DOI: 10.1016/j.celrep.2016.03.042. http://www.nature.com/news/hiv-overcomes-crispr-gene-editing-attack-1.19712
- Healy M. 2016. Could a gene-editing tweak make pigs organ donors for ailing humans? http://www.latimes.com/science/sciencenow/la-sci-sn-gene-editing-pigs-organs-20160316-story.html
Imagen tomada de Crédito. An anti-CRISPR for gene editing. 2016. https://phys.org/news/2016-12-anti-crispr-gene.html. Pawluk A. et al. 2016. Naturally occurring off-switches for CRISPR-Cas9. Cell 167. http://www.cell.com/cell/fulltext/S0092-8674(16)31589-6 , DOI: 10.1016/j.cell.2016.11.017
 PIEL-L Latinoamericana Publicacion periodica en dermatologia | Fundada en 1998
PIEL-L Latinoamericana Publicacion periodica en dermatologia | Fundada en 1998