La reacción en cadena de la polimerasa (Polymerase Chain Reaction: PCR de sus siglas en inglés) no es más que un ensayo enzimático mediado por la acción de la enzima DNA polimerasa (DNApol), que permite la amplificación in vitro, en ciclos repetidos de reacción de secuencias específicas de DNA (llamadas también templado o secuencia blanco). Los elementos involucrados en tal reacción son los siguientes: a. DNA, obtenido de cualquier organismo eucariota ej. levadura, hongo, humano, entre otros, o procariota ej. bacterias, virus; b. una secuencia de oligonucleótidos iniciadores, llamados cebadores o primers, definidos por la complementariedad en su secuencia de bases a una porción terminal de la secuencia de DNA blanco; c. deoxiribonucleótidos trifosfato [dNTP: deoxiadenina trifosfato (dATP); deoxicitidina trifosfato (dCTP); deoxitimidina trifosfato (dTTP); y deoxiguanidina trifosfato (dGTP)], que son las unidades de construcción de la secuencia del DNA; y d. la enzima DNApol cuya temperatura óptima es de 37 °C, o variantes como la Tag DNApol, aislada de la bacteria termófila Thermophilus aquaticus, con una temperatura óptima sobre 70 °C.1,2
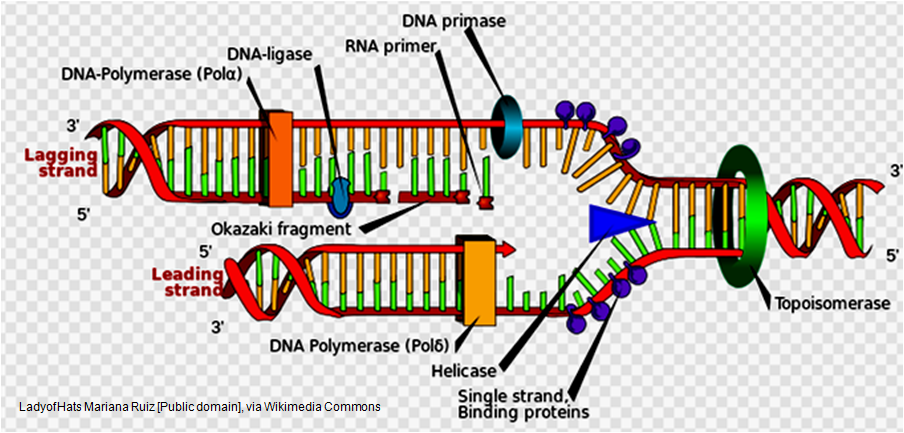
El ensayo está basado en el proceso de replicación o síntesis de DNA de un organismo que ocurre durante la división celular, donde a través de un proceso mediado por un complejo enzimático la doble hélice del DNA nativo es abierta en un sitio particular llamado origen de replicación generando parcialmente dos cadenas sencillas (fusión o denaturalización); dado que las cadenas que forman la doble hélice del DNA son anti paralelas las mismas tienen diferente orientación, una en sentido 5’-3’ y la otra en sentido 3’-5’, de acuerdo a la direccionalidad que une a los respectivos nucleótidos (Ver figura). Una enzima particular del complejo enzimático media la síntesis de un RNA iniciador (cebador o primer) en sentido 5’-3’, que promueve a su vez el inicio de la síntesis de DNA por parte de la enzima DNApol sobre la cadena molde, en dirección 5’-3’, mediante la incorporación de dNTP existentes en el medio de acuerdo al principio de complementariedad de bases. Una vez que se completa el proceso, el RNA iniciador es eliminado y reemplazado por DNA por acción enzimática.3 El proceso de replicación por parte de la DNApol tiene un óptimo de temperatura de 37 °C.
In vitro, el proceso de denaturalización del DNA total o de una secuencia particular de DNA puede llevarse a cabo bajo dos condiciones diferentes: a. incremento de pH mediante alcalinización de la solución de DNA, por ej. utilizando hidróxido de sodio, y b. incremento de temperatura mediante el calentamiento de la solución de DNA sobre 90 °C. Cualquiera de estas dos condiciones en un ensayo de amplificación de DNA por PCR impide la utilización de la DNApol aislada de varios organismos, ya que el pH y temperatura óptima de las mismas es de 7 y 37 °C respectivamente, de allí la importancia de contar con una enzima como la Taq-DNApol cuyo óptimo de temperatura se encuentra sobre los 70 °C.
La transición entre el estado nativo del DNA (doble hélice) y su denaturalización ocurre en un rango de temperatura, permitiendo definir la temperatura de fusión del DNA o Tm como la tempera a la cual la mitad de la secuencia de DNA en estudio se encuentra en cadena sencilla y la otra mitad en su estado de doble cadena. El valor de Tm caracteriza una muestra de DNA y aunque depende de varios factores, el contenido de Guanina-Citocina (G-C) es determinante, mayor contenido de G-C en una secuencia de DNA mostrará mayor Tm en comparación con una secuencia rica en Adenina-Timina (A-T).4
En los ciclos de temperatura diseñados en un ensayo de PCR, el Tm de los cebadores o primers (Tmp) es de suma importancia, ya que la selección de la temperatura de anillamiento o hibridación de los cebadores dependerá de la longitud y composición de los mismos. Una consideración importante es la selección de un Tm igual o muy parecido entre los cebadores. La especificidad del ensayo esta en relación directa con el rango de Tmp, mientras mayor es dicha temperatura (ej. 60 °C) mayor será la especificidad de anillamiento de los cebadores.5
La selección adecuada de la temperatura de anillamiento de los oligonucleótidos cebadores en un ensayo de PCR, es determinante en la especificidad y rendimiento del ensayo. De allí la recomendación de no olvidar tomar las previsiones al respecto, ya que no siempre las temperaturas definidas en un kit o en la literatura son las más apropiadas para el ensayo particular que Ud. quiere llevar a cabo, la variabilidad genética existe y el éxito de su ensayo puede estar relacionado con el diseño de la secuencia de sus cebadores y la selección de la temperatura de anillamiento en su ensayo.
Referencias
- Zagal R. (2012, abril 15). PCR o Reacción en Cadena de la Polimerasa [YouTube]. Recuperado de https://www.youtube.com/watch?v=V9PtQlp-e7g
- CanalDivulgacion IQOG-CSIC. (2014, enero 2). PCR: Reacción en Cadena de la Polimerasa (IQOG-CSIC) [YouTube]. Recuperado de https://www.youtube.com/watch?v=TalHTjA5gKU
- Yourgenome.org [sede Web].Cambridge, UK: YG; 2017 [acceso 14 de julio 2017]. De YG.What is DNA replication? Disponible en: http://www.yourgenome.org/facts/what-is-dna-replication
- Propanona.blogspot.com [sede Web]. Mountain View: Blogger; 2017 [acceso 14 de julio 2017]. De Blogger. Desnaturalización o fusión del ADN. Disponible en: http://propanona.blogspot.com/2014/08/desnaturalizacion-o-fusion-del-adn.html
- Pinzón AM, editor. Introducción al diseño “in silico” de primers. [Internet]. Bogotá: Laboratorio de Micología y Fitopatología de la Universidad de los Andes; 2007 [citado 14 julio 2017].Disponible en: http://bioinf.ibun.unal.edu.co/cbib/estudiantes/1-07/primerDesign.pdf
 PIEL-L Latinoamericana Publicacion periodica en dermatologia | Fundada en 1998
PIEL-L Latinoamericana Publicacion periodica en dermatologia | Fundada en 1998